ໃນຂະບວນການຂອງການສຶກສາທາງພັນທຸກໍາ, ພວກເຮົາມັກຈະພົບກັບຕົວຢ່າງ RNA ທີ່ບໍ່ພຽງພໍ, ສໍາລັບການຍົກຕົວຢ່າງ, ສໍາລັບການສຶກສາ tumors ທາງປາກຂະຫນາດນ້ອຍ, ເຖິງແມ່ນວ່າຕົວຢ່າງຈຸລັງດຽວ, ແລະຕົວຢ່າງຂອງການກາຍພັນຂອງ gene ສະເພາະທີ່ໄດ້ຖືກຖ່າຍທອດໃນລະດັບຕ່ໍາຫຼາຍໃນຈຸລັງຂອງມະນຸດ.ແນ່ນອນ, ສໍາລັບການທົດສອບ COVID-19, ຖ້າ swabs ບໍ່ຢູ່ໃນສະຖານທີ່ທີ່ເຫມາະສົມຫຼືເວລາບໍ່ພຽງພໍໃນລະຫວ່າງການເກັບຕົວຢ່າງ, ຂະຫນາດຕົວຢ່າງຈະຕໍ່າຫຼາຍ, ນັ້ນແມ່ນເຫດຜົນທີ່ຄະນະກໍາມະການສຸຂະພາບແລະການວາງແຜນຄອບຄົວໄດ້ອອກມາສອງມື້ກ່ອນຫນ້ານີ້ແລະ. ໄດ້ຜ່ານການທົດສອບ, ແລະຖ້າຫາກວ່າຕົວຢ່າງອາຊິດ nucleic ບໍ່ໄດ້ເອົາຫົກຕົວຢ່າງ, ທ່ານສາມາດລາຍງານມັນໄດ້.
ຄວາມອ່ອນໄຫວຂອງ reagent ແມ່ນສໍາຄັນເພາະວ່າພວກເຮົາມີບັນຫານີ້ຫຼືບັນຫານັ້ນ, ດັ່ງນັ້ນພວກເຮົາສາມາດເຮັດແນວໃດເພື່ອປັບປຸງຄວາມອ່ອນໄຫວຂອງ RT-PCR?
ກ່ອນທີ່ພວກເຮົາຈະປຶກສາຫາລືກ່ຽວກັບການແກ້ໄຂທີ່ເປັນໄປໄດ້, ໃຫ້ພວກເຮົາກ່າວເຖິງສອງອາການແຊກຊ້ອນໃຫຍ່ກັບສະຖານະການທີ່ພວກເຮົາພຽງແຕ່ໄດ້ກ່າວມາ.
ກ່ອນອື່ນ ໝົດ, ພວກເຮົາກັງວົນກ່ຽວກັບການສູນເສຍ RNA ເມື່ອພວກເຮົາມີພຽງແຕ່ສອງສາມຈຸລັງໃນຕົວຢ່າງຂອງພວກເຮົາ.ຖ້າວິທີການແຍກແລະການທໍາຄວາມສະອາດແບບດັ້ງເດີມຖືກນໍາໃຊ້, ເຊັ່ນວິທີການຖັນຫຼືວິທີການ precipitation ອາຊິດ nucleic, ມີຄວາມເປັນໄປໄດ້ສູງທີ່ຕົວຢ່າງຈໍານວນຫນ້ອຍຈະສູນເສຍ.ການແກ້ໄຂຫນຶ່ງແມ່ນການເພີ່ມໂມເລກຸນຂອງຜູ້ໃຫ້ບໍລິການ, ເຊັ່ນ tRNA, ແຕ່ເຖິງແມ່ນແລ້ວ, ບໍ່ມີການຮັບປະກັນວ່າການທົດລອງການຟື້ນຟູຂອງພວກເຮົາແມ່ນດີ.
ດັ່ງນັ້ນວິທີທີ່ດີກວ່າແມ່ນຫຍັງ?ທາງເລືອກທີ່ດີສໍາລັບຈຸລັງວັດທະນະທໍາຫຼືຕົວຢ່າງ microanatomical ແມ່ນການນໍາໃຊ້ lysis ໂດຍກົງ.
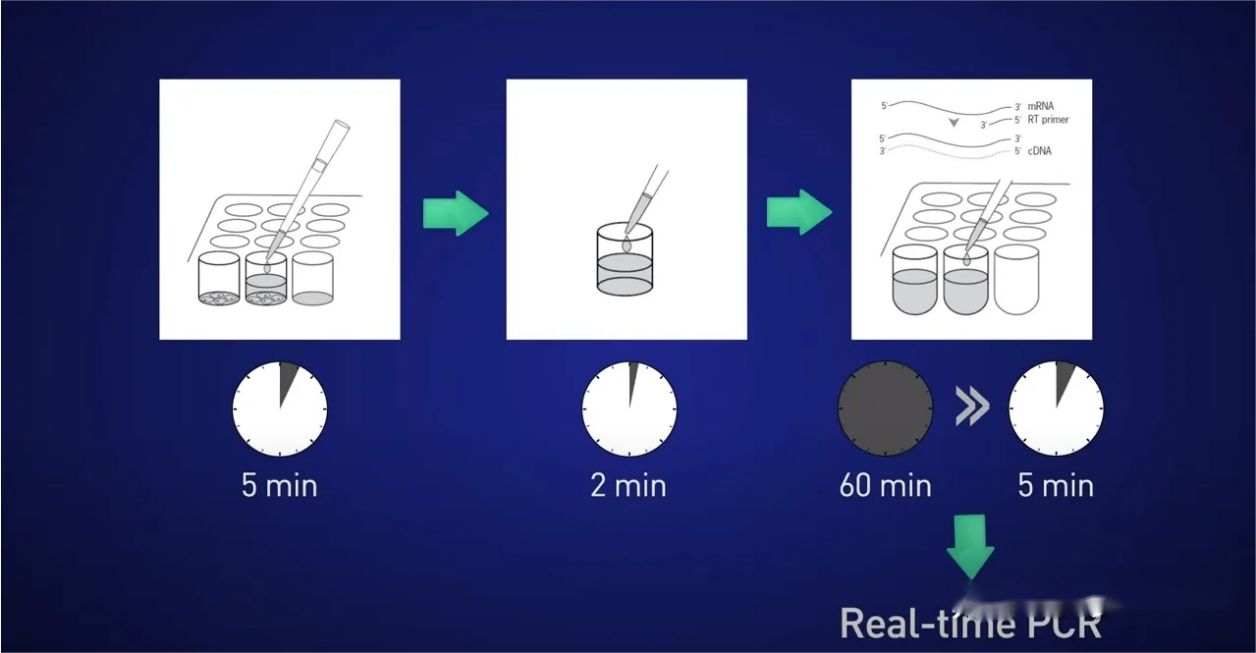
ແນວຄວາມຄິດຄືການແບ່ງຈຸລັງເປັນເວລາ 5 ນາທີ, ປ່ອຍ RNA ເຂົ້າໄປໃນການແກ້ໄຂ, ຫຼັງຈາກນັ້ນຢຸດຕິກິຣິຍາສໍາລັບ 2 ນາທີ, ຫຼັງຈາກນັ້ນຕື່ມ lysate ໂດຍກົງກັບປະຕິກິລິຍາ reverse transcription ເພື່ອບໍ່ໃຫ້ RNA ສູນເສຍ, ແລະສຸດທ້າຍເອົາ cDNA ຜົນໄດ້ຮັບໂດຍກົງ. ເຂົ້າໄປໃນປະຕິກິລິຍາໃນເວລາຈິງ.
ແຕ່ຈະເປັນແນວໃດຖ້າ, ເນື່ອງຈາກຈຸດເລີ່ມຕົ້ນທີ່ຈໍາກັດຫຼືຈໍານວນການສະແດງອອກຂອງ gene ເປົ້າຫມາຍເລັກນ້ອຍ, ພວກເຮົາສາມາດ recycle RNA ທັງຫມົດແລະຍັງບໍ່ມີແມ່ແບບພຽງພໍເພື່ອໃຫ້ໄດ້ສັນຍານໃນເວລາທີ່ແທ້ຈິງ?
ໃນກໍລະນີນີ້, ຂັ້ນຕອນການຂະຫຍາຍເບື້ອງຕົ້ນສາມາດເປັນປະໂຫຍດຫຼາຍ.
ຕໍ່ໄປນີ້ແມ່ນໂຄງການທີ່ຈະເພີ່ມຄວາມອ່ອນໄຫວຫຼັງຈາກການຖອດຖອນຄືນ.ກ່ອນທີ່ຈະເລີ່ມຕົ້ນ, ພວກເຮົາຈໍາເປັນຕ້ອງຖາມລຸ່ມນ້ໍາວ່າເປົ້າຫມາຍໃດທີ່ພວກເຮົາສົນໃຈ, ເພື່ອອອກແບບ primers ສະເພາະສໍາລັບເປົ້າຫມາຍເຫຼົ່ານີ້ສໍາລັບການຂະຫຍາຍກ່ອນ.
ນີ້ສາມາດເຮັດໄດ້ໂດຍການສ້າງ primer ປະສົມທີ່ມີ primers ສູງເຖິງ 100 ຄູ່ແລະຮອບວຽນຕິກິຣິຍາຂອງ 10 ຫາ 14 ເທື່ອ.ດັ່ງນັ້ນ, Master Mix ທີ່ຖືກອອກແບບໂດຍສະເພາະສໍາລັບຄວາມຕ້ອງການນີ້ແມ່ນຈໍາເປັນເພື່ອຂະຫຍາຍ cDNA ທີ່ໄດ້ຮັບກ່ອນ.
ເຫດຜົນສໍາລັບການກໍານົດຈໍານວນຂອງຮອບວຽນລະຫວ່າງ 10 ແລະ 14 ແມ່ນວ່າຈໍານວນຮອບວຽນຈໍາກັດນີ້ຮັບປະກັນຄວາມສຸ່ມລະຫວ່າງເປົ້າຫມາຍຕ່າງໆ, ເຊິ່ງເປັນສິ່ງສໍາຄັນສໍາລັບນັກຄົ້ນຄວ້າທີ່ຕ້ອງການຂໍ້ມູນໂມເລກຸນປະລິມານ.
ຫຼັງຈາກການຂະຫຍາຍທາງສ່ວນຫນ້າ, ພວກເຮົາສາມາດໄດ້ຮັບຈໍານວນຂະຫນາດໃຫຍ່ຂອງ cDNA, ດັ່ງນັ້ນຄວາມອ່ອນໄຫວໃນການກວດສອບຢູ່ດ້ານຫລັງແມ່ນການປັບປຸງຢ່າງຫຼວງຫຼາຍ, ແລະພວກເຮົາຍັງສາມາດເຈືອຈາງຕົວຢ່າງແລະປະຕິບັດປະຕິກິລິຍາ PCR ໃນເວລາທີ່ແທ້ຈິງຫຼາຍເພື່ອລົບລ້າງຄວາມຜິດພາດທີ່ອາດຈະເປັນໄປໄດ້.
ເວລາປະກາດ: 11-04-2023